Bioinformatik (Prof. Helms)
21. Oktober, 2022 2023-04-13 1:33Bioinformatik (Prof. Helms)
Über uns
Die Arbeitsgruppe Helms verwendet numerische Simulationen und statistische Methoden um biomolekulare Wechselwirkungen und Regelkreise in der Zelle zu verstehen. Wichtige Themen sind derzeit die Modellierung von Protein-Protein- und Protein-DNA-Kontakten, die zentrale Steuerungsaufgaben in allen wichtigen biologischen Prozessen einnehmen. Daneben arbeiten wir auf dem Gebiet der Membranbioinformatik daran, strukturelle und funktionelle Merkmale von integralen Membranproteinen aus deren Proteinsequenzen abzuleiten. Ein besonderer Fokus liegt darauf, die Brücke von dem Verständnis einzelner molekularer Interaktionen zu den größeren Zusammenhängen in biologischen Prozessen zu schlagen. Dadurch kann die Entstehung von Krankheiten auf molekularer Ebene verstanden werden.
Gruppenleiter

Prof. Dr. Volkhard Helms
Volkhard Helms ist seit 2003 Professor für Bioinformatik am Zentrum für Bioinformatik Saar und Mitglied der Zentrumsleitung. Er studierte Physik in Freiburg und München und schloß 1996 seine Dissertation am Europäischen Laboratorium für Molekularbiologie in Heidelberg ab. Im Anschluß an eine Post-Doc-Phase an der University of California, San Diego, übernahm er die Leitung einer selbständigen Nachwuchsgruppe für theoretische Biophysik am Max-Planck-Institut für Biophysik in Frankfurt. 2001 wurde er als EMBO Young Investigator ausgezeichnet.
"Die Gruppe möchte über die Modellierung einzelner biomolekularer Wechselwirkungen und Regelvorgänge zum umfassenden Verständnis zellbiologischer Prozesse gelangen."
Unsere Projekte
Funktionelle Zuordnung von Transmembranproteinen
Etwa ein Viertel aller Eiweißmoleküle werden in die Zellmembran oder in die Membranen von Zellkompartimenten integriert. Dort vermitteln sie den Transport von kleinen Molekülen durch die Membran und leiten die Signale von Botenstoffen, die an die Zelle andocken, in das Zellinnere weiter. Wir interessieren uns vor allem dafür, die bisher weitgehend unbekannte Zuordnung aufzuklären, welche Membrantransporter welche Substratmoleküle durch die Membran transportieren. Dazu entwickeln wir neuartige statistische Methoden sowie eine Softwareumgebung zur Modellierung einer virtuellen Zellmembran.
Beeinflussung von Protein-Protein-Wechselwirkungen
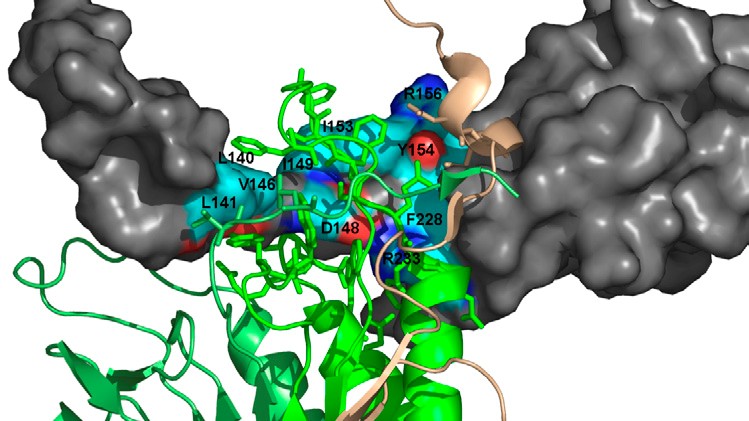
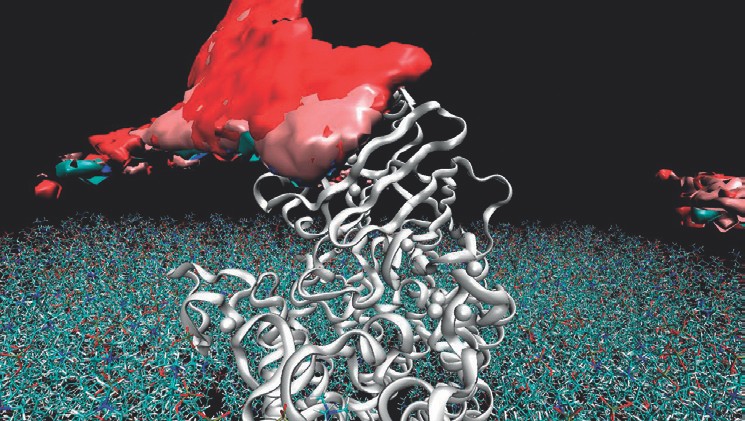
Die meisten Proteine interagieren in Zellen ständig mit anderen Proteinen. Wir verwenden aufwändige Computersimulationen, um diese Assoziationsprozesse im atomarem Detail entschlüsseln zu können. Wir fanden heraus, dass das Lösungsmittel Wasser zwischen den Proteinen eine verstärkende Rolle bei der Zusammenlagerung spielt. Basierend auf diesen Erkenntnissen beschäftigen wir uns nun damit, wie kleine Ligandenmoleküle diese Protein-Wechselwirkungen blockieren oder, umgekehrt, verstärken können. Diese Strategie ermöglicht es, neue Therapien z.B. bei der Bekämpfung von Krebs zu entwickeln.