Bioinformatik (Prof. Lenhof)
10. Oktober, 2022 2023-11-23 13:45Bioinformatik (Prof. Lenhof)
Über uns
Die Arbeitsgruppe Lenhof entwickelt neue bioinformatische Methoden und Software für die Aufklärung der Mechanismen von pathogenen Prozessen und für die Diagnose, Prognose und die Therapie von verschiedenen Krankheiten.

Gruppenleiter

Prof. Dr. Hans-Peter Lenhof
Hans-Peter Lenhof studierte Mathematik und Chemie an der Universität des Saarlandes (Diplom in Mathematik 1989). 1993 schloss er seine Dissertation im Fach Informatik an der Universität des Saarlandes ab. Von 1993 bis 1999 forschte er als Post-Doc am Max-Planck-Institut für Informatik in Saarbrücken. 1999 habilitierte er im Fach Informatik an der Universität des Saarlandes und übernahm eine Forschungsgruppenleiterstelle (C3) am MPI für Informatik. Seit 2000 leitet er einen Lehrstuhl für Bioinformatik an der Universität des Saarlandes.
"Unsere Forschung konzentriert sich auf die Entwicklung neuer bioinformatischer Ansätze und Methoden zum Studium und zur Aufklärung der Mechanismen der Tumorentstehung und -entwicklung mit dem Ziel, die Diagnose, Prognose und Therapie von Tumoren zu verbessern."
Unsere Projekte
Therapie
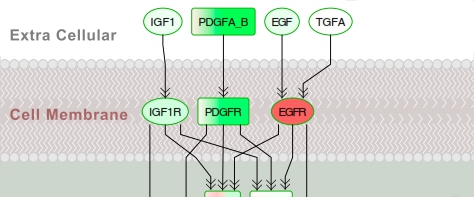
Krebs ist eine heterogene Gruppe von Krankheiten. Selbst Krebserkrankungen desselben (Sub-)Typs können sich stark in ihrem Geno- und Phänotyp unterscheiden. Aufgrund dieser Heterogenität benötigt jeder Tumorpatient eine personalisierte Therapie. Heute stehen mehr als 200 Krebsmedikamente zur Verfügung. Die Herausforderung besteht jedoch darin, das beste Krebsmedikament oder die beste Medikamentenkombination für einen bestimmten Tumor zu finden.
Unsere Arbeitsgruppe entwickelt neue Ansätze zur Unterstützung der Optimierung von Tumortherapien. Insbesondere fokussieren wir uns dabei auf KI/ML-Methoden für die Vorhersage wirksamer Therapien.
Diagnose und Prognose
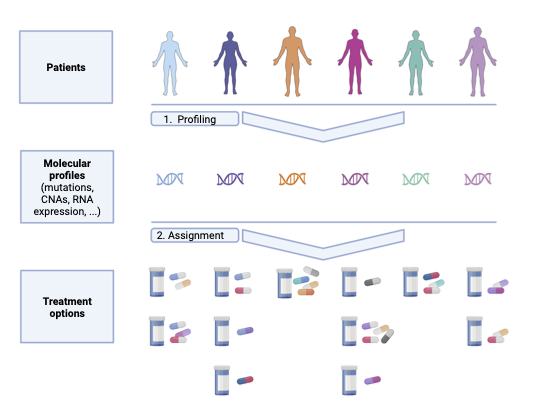
In enger Zusammenarbeit mit den Arbeitsgruppen von Andreas Keller (Klinische Bioinformatik, Universität des Saarlandes und HIPS) und Eckart Meese (Humangenetik, Universität des Saarlandes) erforschen wir neue diagnostische Ansätze, die auf umfangreichen Antikörper- und miRNA-Profilen basieren. In ersten Machbarkeitsstudien wurden Autoantikörper- und miRNA-Profile in Blutseren von Patienten mit verschiedenen Erkrankungen und einer Vergleichsgruppe bestimmt. Klassifikationsverfahren, die auf diesen Profilen trainiert und evaluiert wurden, erreichten für einige Krankheiten, unter anderem Meningiome, Gliome, Lungentumore, Melanome und Multiple Sklerose (MS), eine hohe Sensitivität und Spezifität. Unsere Ergebnisse zeigen deutlich, dass Antikörper- und miRNA-Profile großes diagnostisches Potential besitzen.
Aufklärung der Mechanismen von pathogenen Prozessen
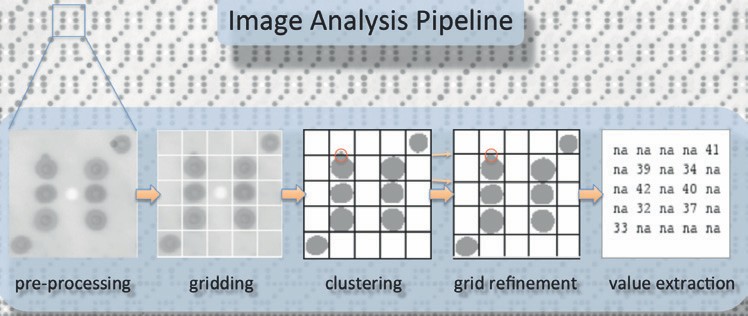
Komplexe biologische Prozesse können als Ketten bzw. Netzwerke von einzelnen Reaktionen aufgefasst werden und können daher als Graphen modelliert werden, wobei die Knoten und Kanten der Graphen die beteiligten Biomoleküle und deren Reaktionen und Interaktionen repräsentieren. Wir entwickeln neue Graph-basierte Ansätze für die Analyse und Visualisierung von biologischen Prozessen. Unser besonderes Interesse gilt hierbei der Entwicklung neuer Verfahren zur Detektion und Analyse von deregulierten regulatorischen Prozessen und Signalkaskaden, welche charakteristisch für bestimmte pathogene Prozesse und Krankheiten, wie z.B. Krebs sind.