Forschungsthemen im Bereich der Bioinformatik
27. März, 2023 2023-04-13 1:33Forschungsthemen im Bereich der Bioinformatik
Vom Genom zum Medikament
Die Bioinformatik spielte eine entscheidende Rolle beim bedeutendsten wissenschaftlichen Ereignis der letzten Jahre: Die Sequenzierung des menschlichen Genoms. Nachdem die Sequenz vorliegt, folgt jetzt die Entschlüsselung des Genoms.

Der Vorgang der Entschlüsselung wird als Annotation bezeichnet. Darunter versteht man die Suche nach den Bauplänen (Genen) für die einzelnen Bausteine (zum Beispiel Proteine) des menschlichen Köpers sowie die Aufklärung ihrer Funktion und Struktur.
Die Funktion eines einzelnen Bausteins kann nur unter Berücksichtigung des Zusammenspiels mit anderen Bausteinen erforscht werden. Diese Wechselwirkungen werden in sogenannten metabolischen und regulatorischen Netzwerken dargestellt, die unter anderem die Simulation des Stoffwechsels im Computer ermöglichen. Mit Bioinformatikmethoden sucht man nach den Ursachen genetisch bedingter Krankheiten, um neue Wege für Therapien zu finden.
Medikamente (Wirkstoffe) wirken durch Beeinflussung bestimmter Bausteine (Proteine) des Stoffwechsels. Mit Beginn der Entschlüsselung des menschlichen Genoms wird es mit Hilfe der Bioinformatik möglich, viele neue und für eine Behandlung geeignetere Proteine als Angriffspunkte (Targets) zu finden.
Die Bioinformatik trägt auch wesentlich zur Entwicklung neuer Wirkstoffe bei: In großen Wirkstoff-Datenbanken wird nach geeigneten Kandidaten gesucht (Screening, Docking), die effektiv sind, möglichst wenig unerwünschte Nebenwirkungen haben und zudem in der Lage sind, den Zielort im Körper zu erreichen (Überwindung biologischer Barrieren).
Die Optimierung bekannter Therapien wird durch Verfahren der Bioinformatik unterstützt: Durch den Vergleich ganzer Genome verschiedener Individuen können Unterschiede (z.B. SNPs) gefunden werden, die eine Individualisierung von Therapien ermöglicht.
Virale Infekte stellen eine große Herausforderung in Wirkstoffdesign und Therapie dar. Auf Grund der Tatsache, dass Viren wie HIV eine genomische Variabilität besitzen, kann es vorkommen, dass virale Mutationen auftreten, die eine Resistenz gegen die verabreichten Medikamente aufweisen.
Aus diesem Grund sind Ärzte relativ häufig mit dem Problem konfrontiert, einen individuellen Therapieansatz für jeden Patienten mit einer bestimmten Erregervariante zu finden. Um bessere therapeutische Strategien zu wählen wurden bioinformatische Methoden entwickelt, die es erlauben, den Zusammenhang zwischen viraler Mutation und Wirkstoff-Resistenz besser zu verstehen.
Sequenzierung des Genoms
Das menschliche Genom besteht aus 46 langen DNA-Molekülen (Chromosomen), die im Zellkern enthalten sind. Die Chromosomen enthalten die komprimierte Erbinformation.
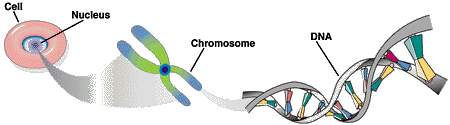
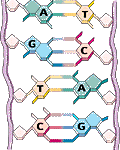
Jedes Chromosom besteht aus einem DNA-Doppelstrang: Zwei Einzelstränge winden sich in Form einer Doppelhelix umeinander. Jeder DNA-Einzelstrang besteht aus einer Abfolge von Bausteinen (Nukleotiden), die durch die jeweils enthaltene Base charakterisiert sind.
Es gibt insgesamt vier verschiedene Bausteine, die mit den vier Buchstaben A, C, G und T bezeichnet werden. Je zwei Nukleotide (A+T, G+C) sind komplementär, das heißt sie binden aneinander. Auf diese Weise bildet sich der Doppelstrang. Von den beiden Einzelsträngen wird daher nur einer benötigt, da der jeweils andere aufgrund der Basenkomplementarität eindeutig definiert ist. Das menschliche Genom kann daher als lange Zeichenkette bestehend aus den vier Buchstaben A, T, G und C dargestellt werden.
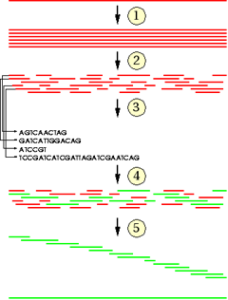
Ziel der Sequenzierung ist es, die Erbinformation, die in den Chromosomen enthalten ist, in Form einer langen Textzeile zu erhalten. Die verwendeten Sequenziermaschinen sind jedoch nicht in der Lage, das gesamte Genom in einem Schritt zu lesen. Aus diesem Grund muss das Genom in kleine Stücke zerteilt werden, deren Buchstabenfolge gelesen werden kann. Um diese Einzelteile später wieder zusammensetzen zu können, müssen die einzelnen Bruchstücke überlappen.
Dies kann man dadurch erreichen, indem man viele Kopien eines DNA-Einzelstranges erzeugt und jede Kopie zufällig zerstückelt (zum Beispiel mit Ultraschall, hoher Druck). Die vielen Einzelabschnitte werden anschließend von Maschinen gelesen.
Die Gesamtabfolge der Bausteine wird mit Hilfe der Überlappungsinformationen gewonnen: Schrittweise werden Einzelstücke gesucht, deren Enden überlappen. Diese überlappenden Abschnitte werden in der entsprechenden Reihenfolge zusammengefügt. Diesen Vorgang nennt man Assemblierung.
Algorithmen zur Assemblierung müssen sehr effizient sein, da die Zahl der Bruchstücke und damit die Zahl der paarweisen Vergleiche auf mögliche Überlappungen sehr hoch ist. Zudem müssen die Verfahren mit Problemen wie z.B. mehrfach im Genom auftretenden Teilsequenzen oder Lesefehlern bei den Bruchstücken fertig werden können.
Annotation der Gene
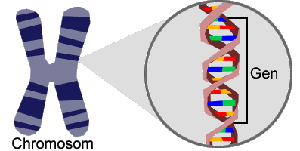
Die Chromosomen beinhalten das Genom eines Lebewesens. Das Genom enthält Abschnitte (Gene), die die Baupläne für Proteine und andere Bausteine kodieren. Der Anteil an genkodierender Sequenz im Gesamtgenom ist verhältnismäßig gering.
Ist das Genom sequenziert, müssen also zunächst die Gene gesucht und erkannt werden. In diesem Schritt, der so genannten Annotation, kommen Verfahren der Bioinformatik wie spezielle Mustererkennung und Sequenzalignment zum Einsatz.
Struktur und Funktion von Genprodukten
Hat man ein Gen gefunden, so will man die Struktur und die Funktion des Bausteins aufklären, der durch das Gen kodiert wird. Die Sequenz eines Gens bestimmt die räumliche Struktur des Bausteins.
Diese dreidimensionale Form hat wiederum Einfluss darauf, welche Aufgabe der betreffende Baustein im Körper wahrnimmt. Die Bioinformatik entwickelt sowohl Algorithmen, die eine Vorhersage der räumlichen Struktur eines Bausteins ermöglichen, als auch Verfahren zur Funktionsanalyse.

Da die Sequenz die Struktur eines Bausteins bestimmt, bietet es sich an, in Datenbanken nach ähnlichen Sequenzen zu suchen, um das Wissen über die Funktion schon bekannter Bausteine auf neu sequenzierte Gene zu übertragen (Sequenzalignment). Auch durch den Vergleich der räumlichen Struktur kann man die Funktion von Bausteinen bestimmen.
Ein Erfolg versprechender Ansatz zur Funktionsaufklärung ist die Verwendung so genannter DNA-Chips bzw. der mit Hilfe dieser Technik erzeugten Messdaten: DNA-Chips erlauben es, Informationen darüber zu erhalten, welche Proteine zu einem bestimmten Zeitpunkt in der Zelle aktiv sind und welche nicht. Auf diese Weise kann man Informationen über die wahrscheinliche Funktion von Proteinen bestimmen.

Metabolische Netzwerke
Nach der Aufklärung der Funktion einzelner Proteine ist es von besonderem Interesse, die metabolischen Pfade (Metabolic Pathways) eines Organismus zu untersuchen.
Als metabolischen Pfad bezeichnet man eine Abfolge von Reaktionen, die zusammen für die Umsetzung eines Stoffes in einen anderen sorgen. Die einzelnen Reaktionen sind dabei im Wesentlichen durch das katalysierende (die Reaktion beschleunigende) Enzym charakterisiert.
Wichtige Beispiele für metabolische Pfade sind der Citratzyklus oder die Glykolyse. Die Gesamtheit aller Pfade bildet ein metabolisches Netzwerk.
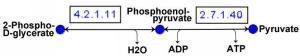
Neben den metabolischen gibt es die ebenso wichtigen regulatorischen Pfade, die die Kommunikation zwischen den Zellen und die gesamte Steuerung der Vorgäge im Organismus organisieren. Mit Hilfe verschiedenster Signale und Kommunikationsmechanismen können die Vorgänge im Organismus gezielt gesteuert werden. Alle regulatorischen Pfade zusammen bilden das regulatorische Netzwerk eines Organismus.
Um sowohl metabolische als auch regulatorische Netzwerke mit Hilfe des Computers analysieren zu können, benötigt man entsprechende Datenmodelle. Ziel ist es, ein geeignetes Modell zu finden, das beide Netzwerkarten repräsentieren kann.
Die Netzwerkrepräsentationen bilden die Grundlage zur Entwicklung neuer Werkzeuge, die zum Beispiel für Target-Identifizierung, Wirkstoffdesign oder die Aufklärung der Ursachen genetisch bedingter Krankheiten benötigt werden.
In der Grundlagenforschung können diese Netzwerke für den Vergleich von Stoffwechselabläufen verschiedener Organismen herangezogen werden. Zum Beispiel können Informationen über den Metabolismus eines Organismus verwendet werden, um das neu sequenzierte Genom (und damit die Stoffwechselwege) eines anderen Organismus zu verstehen.
Target-Identifizierung
Mit Hilfe der der metabolischen und regulatorischen Netzwerke können die Wissenschaftler daran gehen, den Stoffwechsel im menschlichen Organismus nachzuvollziehen. Besonders interessiert sind sie dabei an Störungen der normalen Abläufe, da diese häufig Ursache für Erkrankungen sind.
Weiß man, wo die Ursache einer Krankheit liegt, kann man zum Beispiel mit Hilfe der metabolischen Netzwerke eine genauere Analyse vornehmen und “Angriffspunkte” (daher “Target”-Identifizierung) für eine mögliche Behandlung suchen. Die gesuchten Targets sind in aller Regel Enzyme, die bestimmte Reaktionen im Stoffwechsel steuern.
Hat man ein Enzym als “Übeltäter” ermittelt, kann man versuchen, mit bestimmten Wirkstoffen Einfluss auf die Aktivität dieses Enzyms zu nehmen, um so die Ursache der Krankheit zu beheben. Die Verwendung der zuvor modellierten Netzwerke ermöglicht dabei auch die Analyse möglicher Nebenwirkungen auf andere Prozesse im Körper, die durch eine Behandlung nicht beeinflusst werden dürfen.
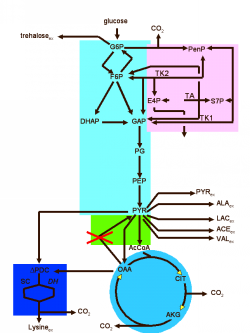
Wirkstoffdesign
Die Identifizierung eines Targets allein genügt noch nicht, um eine wirksame Behandlung vornehmen zu können – zunächst muss ein geeigneter Wirkstoff entwickelt werden. Ein derartiger Wirkstoff muss das Krankheit auslösende Enzym derart beeinflussen, dass dieses nicht mehr störend auf den Stoffwechsel einwirkt.
Dies kann zum einen dadurch geschehen, dass der Wirkstoff das Enzym in seiner Aktivität hemmt. Falls eine Krankheit dadurch entsteht, dass ein Mangel an einem bestimmten Stoff vorliegt, kann ein Wirkstoff auch so konstruiert werden, dass er die Produktion eines Enzyms anregt, das dann seinerseits die notwendigen (und zuvor nicht oder nur ungenügend vorhandenen) Stoffwechselvorgänge steuert.
Auf der molekularen Ebene bedeutet die Beeinflussung der Enzymaktivität durch einen Wirkstoff das “Andocken” des Stoffes an das Zielprotein (Target), um auf diese Weise seine Wirkung zu entfalten.
Aus diesem Grunde wurden bioinformatische Methoden entwickelt, die das Target virtuell auf Liganden hin screenen, die an das Protein binden und dieses inhibieren können. Eine weitere Möglichkeit besteht darin, andere Proteine zu finden, die die Aktivität des Targets durch Binden und Komplexbildung regulieren. In beiden Fällen ist das Docking die Haupttechnik.
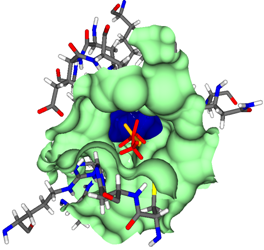
Docking
Proteine kann man sich grob als lange Kette von Aminosäuren vorstellen. Jede Aminosäure besteht aus einer funktionellen Gruppe und dem sogenannten Rest. Die Reste der Aminosäuren bezeichnet man im Protein auch als Seitenketten. Die Kettenbildung erfolgt an der funktionellen Gruppe, an der die einzelnen Proteine eine Peptid-Bindung eingehen. Diese Kettenbildung kann man theoretisch beliebig lang fortführen. Die verschiedenen Aminosäuren werden im Allgemeinen mit einem Code aus 3 Buchstaben abgekürzt. Will man ein Protein beschreiben, so reicht es, diese Folge von Aminosäuren aufzuschreiben.
Die theoretische Bestimmung der Wechselwirkungen zwischen zwei Proteinen bzw. Protein und Ligand, also grob gesagt die Bindung, ist von großem Interesse, denn sie ermöglicht die Verifikation von Hypothesen während des sogenannten Drug-Designs, ohne dabei auf reale Laborexperimente angewiesen zu sein.
Ein immer wieder auftretendes Problem ist das so genannte Protein-Protein- Docking, das folgendermaßen definiert ist:
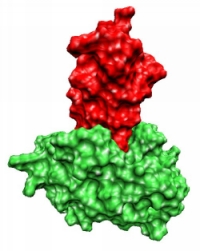
Seien die räumlichen Strukturen der Proteine A und B gegeben, von denen bekannt ist, dass sie einen Komplex AB bilden, dann versteht man unter dem Protein-Protein-Docking die korrekte Vorhersage der Komplexstruktur AB.
Es gibt verschiedene Ansätze für das Protein-Protein- Docking. Der einfachste Ansatz betrachtet Proteine als starre Gebilde. Hier spricht man vom Rigid Body Docking (RBD). Dieser Ansatz geht auf das bekannte Schlüssel- Schloss-Prinzip zurück, das Emil Fischer 1894 vorschlug.
Dieses Modell besagt, dass die zu dockenden Proteine, in seinem Fall Enzym und passendes Substrat, starre Körper sind, die geometrisch komplementäre Regionen besitzen müssen. Zwar sind Proteine keine starren Körper und haben bewegliche Seitenketten, allerdings ergaben Untersuchungen, dass sich der Backbone, also das Grundgerüst bestehend aus den durch Peptidbindung verknüpften funktionellen Gruppen, bei vielen Proteinen während der Komplexbildung kaum verändert.
In der Regel liefert das Docking nicht nur einen Wirkstoffkandidaten, sondern eine große Zahl, die die rein physikalischen Voraussetzungen erfüllen, um mit dem Target zu interagieren. Aus der Menge dieser Kandidaten müssen nun diejenigen ausgewählt werden, die als effektiver Wirkstoff in Frage kommen. Mögliche Probleme können zum Beispiel dadurch entstehen, dass der entwickelte Wirkstoff gar nicht erst den Weg zu seinem Ziel findet, da er im menschlichen Körper einer Vielzahl unterschiedlicher Umgebungen und biologischer Barrieren ausgesetzt ist.
Biologische Barrieren
Sobald ein möglicher Wirkstoff gefunden ist, muss dieser an den Ort des Geschehens gebracht werden. Dafür müssen immer einige biologische Barrieren überwunden werden. Die ersten Barrieren tauchen schon bei der Aufnahme des Stoffes in den Körper auf. Dabei muss die Darmwand oder die Membran der Lungenbläschen durchdrungen werden, bevor der Wirkstoff zersetzt oder ausgeschieden wird.
Außerdem müssen Transportverluste beachtet werden. Selbst wenn man nur in ganz primitive (einzellige) Organismen eingreifen will, sind zumindest Zellmembranen zu überwinden. Zu diesem Zeitpunkt ist insbesondere das praktische Experiment notwendig – ein aufwendiges und teures Verfahren zum Eignungstestung eines Wirkstoffs.
Auf diesem Teilgebiet ist noch viel Forschungsarbeit, insbesondere für die Bioinformatik, nötig, um alternative Methoden zur Verfügung zu stellen, die helfen können, Zeit und Geld zu sparen.

